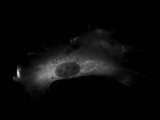
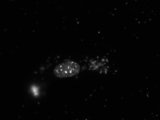
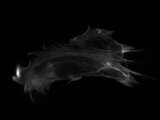
Andrea hat mir vor zwei Tagen über ICQ drei Aufnahmen einer gefärbten Zelle geschickt, die sie mit einem Fluoreszenz-Mikroskop aufgezeichnet hatte. Alles Aufnahmen der selben Probe bei UV-Licht, bei unterschiedlichen Filtern, wenn ich es richtig verstanden habe. Während sie mir noch erklärte, was ich genau man auf den unterschiedlichen Bildern erkennen konnte, bastelte ich die Bilder schnell als eingefärbte Ebenen in GIMP zusammen.
Auf dem Ergebnis aufbauend, hab ich Quick & Dirty ein PHP-Script geschrieben, das diese Aufgabe automatisiert über das Modul php_imagick erledigt – eine Wrapper-Klasse für die bekannte ImageMagick Kommandozeilen-Suite und wohl eines der am schlechteste dokumentierten Module ever. 😉 Einige kurze Beispiele gibt es bei Mikko und im extrem rudimentären Manual. Den Rest muss man sich aus der normalen Doku für die Kommandozeile suchen.
Da die einzelnen Fluoreszenz-Farbstoffe ganz markante Maxima bei einem oder wenigen Wellenlängen haben, wollte ich die Ebenen auch mit der entsprechenden Farbe einfärben. Die Zuordnung Wellenlänge zu RGB Farbe ist allerdings alles andere als trivial. Dabei spielt die Farbwahrnehmung des menschlichen Augen ein große Rolle und schon nach ein paar gelesenen Seiten auf Wikipedia steht man knietief in Farbprofilen und ähnlichem. Das war mir dann doch etwas zu krass.
Dafür habe ich auf der Homepage von Dan Bruton einen Quelltext (in Fortan) gefunden, in dem er über eine einfache Approximation (Geraden) dem sichtbaren Spektrum RGB Farben zugeordnet hat. Das Modell habe ich 1:1 in eine PHP-Funktion übertragen (hoffentlich fehlerfrei) und zur Umrechnung benutzt.
Die Verarbeitungskette habe ich so aufgebaut:
- Die Aufnahmen (Graustufe) als Maske mit einem entsprechend einfarbig gefüllten Bild mit Imagick::COMPOSITE_MULTIPLY verbinden.
- Die Ergebnisse mit Imagick::COMPOSITE_PLUS vor einem schwarzen Hintergrund zusammenfügen.
Meinen ersten Ansatz, die Aufnahmen als Alphachannel zu benutzen, konnte ich mit der spärlichen Doku bisher nicht umsetzen. Das Ergebnis ist so aber auch recht ansehnlich. Vorschläge zur Optimierung sind sehr willkommen. =)
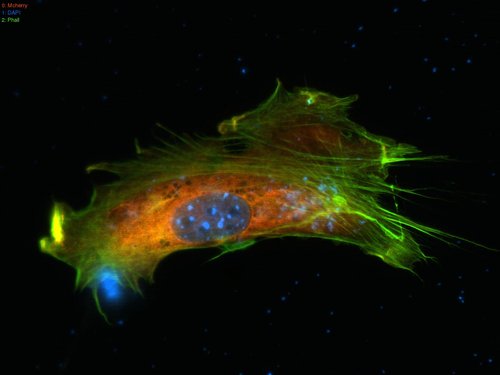
So entsteht aus drei Aufnahmen folgendes Ergebnis:
Hübsch oder? ^^ Die blauen Flecken im Zellkern sind übrigens ein Indiz dafür, dass Irgendwasâ„¢ geklappt hat. Die Veränderung eines Proteins, wenn ich mich recht erinnere.
Das sieht für eine einzelne Ebene im Quellcode dann in etwa so aus:
<?php include_once("class.Wavelength2RGB.php"); $w2rgb = new Wavelength2RGB(); /* calculate color for wavelength */ $color = $w2rgb->wav2rgb(600, 1.0); //600 nm /* Maske laden */ $mask1 = new Imagick($_FILES['mask1']['tmp_name']); /* read width and height */ $o_width = $mask1->getImageWidth(); $o_height = $mask1->getImageHeight(); /* process layer */ $layer1 = new Imagick(); $layer1->newImage( $o_width, $o_height, new ImagickPixel($color)); $layer1->compositeImage($mask1 ,Imagick::COMPOSITE_MULTIPLY ,0 ,0 ,Imagick::CHANNEL_ALL); /* create image */ $output = new Imagick(); $output->newImage($o_width, $o_height, new ImagickPixel("black")); $output->setImageColorspace(1); // 1 - RGBColorspace /* merge layer */ $output->compositeImage($layer1 ,Imagick::COMPOSITE_PLUS ,0 ,0 ,Imagick::CHANNEL_ALL); /* output */ $output->setImageFormat('jpg'); $output->setCompressionQuality(100); $output->writeImage("test.jpg"); ?> |
Inzwischen ist Andrea von ihrer Mentorin auch in die Weiterverarbeitung der Bilder eingeweiht worden. Offensichtlich werden die drei eingesetzten Farbstoffe ganz langweilig einfach den drei Grundkanälen zugeordnet und noch ein wenig an Helligkeit und Kontrast gespielt. Ein Großteil davon geschieht mit zwei Klicks über ein Photoshop-Plugin und somit ist mein Programm nach zwei Tagen schon wieder obsolete. 😉
Auf diesem Webspace kann ich es leider auch nicht laufen lassen, da php_imagick hier (wie wohl bei den meisten Providern) nicht zur Verfügung steht. So ein Ressourcen-Fresser wäre wohl ein zu großes Risiko für die Performance – eigener Server 4tw.

Pingback: Die Macht | caracasa.de
Ja also eigentlich sind nicht die blauen Flecken im Zellkern toll, die sind nämlich immer da, sondern die schwarzen Flecken im schwarzweiss Bild von der Cherry Färbung (erstes Bild).
Das sind nämlich die Nukleoli und wenn man die erkennen kann, dann gelangt das gewünschte Protein in den Zellkern und das wollte ich testen. 🙂
Mist, aber mit Dingern im Kern war ich doch gar nicht so schlecht. 🙂
Sind das dann also die Midi-Chlorianer?